
- 收藏
- 加入书签
基于多视图深度学习的微生物-疾病相关性挖掘?
摘要:微生物与人类健康密切相关,准确挖掘微生物与疾病的相关性对疾病的预防、诊断和治疗具有重要意义。本文提出了一种基于多视图深度学习的微生物-疾病相关性挖掘方法。该方法综合利用微生物的多种属性信息,构建多视图特征表示,并使用深度学习模型学习微生物-疾病相关性。在多个真实数据集上的实验结果表明,该方法能够有效挖掘微生物与疾病的相关性,为疾病的研究提供新的思路。
关键词:微生物;疾病;相关性挖掘;多视图学习;深度学习
引言:
微生物是人体的重要组成部分,与人类健康息息相关。大量研究表明,微生物群落的失衡与多种疾病的发生发展密切相关,如肠道菌群失调与炎症性肠病、肥胖等疾病相关。因此,准确挖掘微生物与疾病的相关性,对疾病的预防、诊断和治疗具有重要意义。传统的相关性分析方法主要基于统计学模型,如相关系数、卡方检验等,但这些方法难以充分利用微生物的多维属性信息。近年来,机器学习尤其是深度学习技术的发展为微生物-疾病相关性挖掘提供了新的思路。本文提出了一种基于多视图深度学习的微生物-疾病相关性挖掘方法,希望为该领域的研究提供新的见解。
1.研究背景与意义
微生物与人体健康密切相关,微生物群落失衡与多种疾病的发生发展存在紧密联系。因此,揭示微生物与疾病之间的相关性对疾病防治具有重要意义。传统微生物-疾病相关性分析主要依赖实验手段,存在成本高、周期长等局限性。高通量测序技术和人工智能的发展为该领域研究提供了新思路。高通量测序使得获取微生物数据更加便捷;而深度学习方法在生物医学领域展现出了巨大潜力,为解决复杂生物学问题提供了新的可能[1]。
本文面向微生物-疾病相关性分析问题,提出了一种基于多视图深度学习的计算方法。该方法从多角度刻画微生物群落特征,结合深度学习技术构建预测模型,较传统方法能够更好地挖掘微生物-疾病相关性中蕴含的复杂模式,具有更强的分析和预测能力。本研究对于加深微生物-疾病相互作用的理解,推动疾病诊断和治疗方法的发展具有重要意义。
2.方法
2.1 传统的相关性分析方法
传统的微生物-疾病相关性分析方法主要包括统计学方法和实验验证方法。统计学方法通过分析微生物组数据和疾病表型数据之间的相关性,鉴定与疾病相关的微生物。常用的统计学方法有差异分析、相关性分析、回归分析等。另一类传统方法是实验验证,通过动物模型或临床试验直接研究微生物与疾病的因果关系。尽管传统方法在微生物-疾病相关性研究中发挥了重要作用,但它们通常只考虑微生物组成信息,忽略了微生物功能、进化关系等其他重要信息,难以全面刻画微生物-疾病相关性。传统方法的分析能力有限,难以应对日益增长的海量微生物组学数据。
2.2 机器学习方法
近年来,机器学习方法在微生物-疾病相关性分析中得到了广泛应用。与传统方法相比,机器学习方法能够从高维微生物组学数据中学习复杂的非线性模式,具有更强的分析和预测能力。一些基于图模型的机器学习方法,能够考虑微生物之间的交互作用,更好地刻画微生物-疾病相关性[2]。尽管取得了一定进展,现有机器学习方法主要基于单一视角构建特征,忽略了多视角信息的整合,难以充分挖掘微生物-疾病相关性。
2.3 多视图学习方法
多视图学习通过整合多个视角的信息,能够更全面、准确地刻画复杂对象。在微生物组学领域,多视图学习方法正受到越来越多的关注。微生物数据具有多个属性维度,能够提供多视图信息。整合多视图信息有助于更好地理解微生物-疾病相关性的内在机制。现有的多视图学习方法主要包括两大类:基于子空间学习的方法和基于多核学习的方法。基于子空间学习的方法通过将不同视图投影到一个公共子空间,实现多视图信息的融合。基于多核学习的方法通过构建多个核函数分别处理不同视图数据,再将多个核函数结合得到最终的预测模型。然而,现有的多视图方法主要是基于浅层模型,难以充分挖掘多视图数据中的深层特征。现有方法忽略了不同视图特征的重要性差异,难以实现最优的特征选择和融合。
3.基于多视图深度学习的疾病相关性挖掘方法
3.1 问题定义
我们将微生物-疾病相关性挖掘问题定义为一个监督学习问题。给定一个微生物组数据集,其中每个样本包含微生物组特征向量和对应的疾病标签,我们的目标是学习一个预测函数,使其能够根据微生物组特征准确预测疾病标签。考虑到微生物组数据的多维属性,我们引入多视图表示,即每个样本可以从多个视图描述其微生物组特征。
3.2 多视图特征构建
为了充分利用微生物组数据的多维属性信息,我们从三个视角构建微生物特征。微生物组成视图从微生物群落的物种组成角度描述微生物组数据,我们使用经过质控和标准化的物种丰度作为特征表示。微生物功能视图从微生物群落的功能角度描述微生物组数据,我们利用基因组注释和功能数据库获得微生物的功能注释信息,并使用功能通路或模块的丰度作为特征表示[3]。微生物进化视图从微生物群落的进化关系角度描述微生物组数据,我们通过构建物种间的进化树或网络,获得微生物之间的进化关系信息,并使用网络嵌入方法学习到低维特征表示。通过这三个视图,我们可以从不同角度描述微生物组数据,获得更全面、多样的特征表示。
3.3 深度学习模型
为了有效学习多视图微生物特征与疾病标签的复杂关系,我们设计了一个端到端的多视图多通道图卷积网络(MVGCN)模型。该模型首先通过图数据增强层去除数据噪声,提高鲁棒性。然后使用多视图GCN编码器,用图卷积网络(GCN)从不同视图学习微生物和疾病节点的嵌入表示。
模型将多视图嵌入堆叠成多通道特征,并用注意力机制自适应地融合不同通道的特征。融合后的特征被输入到CNN中,提取高级特征并整合不同视图的信息。输出层计算微生物-疾病的关联概率,并使用矩阵补全策略预测缺失的关系。整个模型联合优化,通过多视图GCN、注意力机制和CNN的结合,全面地建模微生物-疾病关联预测中的复杂特征交互。
3.4 模型训练与优化
在模型训练过程中,我们使用交叉熵损失函数衡量预测标签与真实标签之间的差异,并使用梯度反向传播算法更新模型参数。为了防止过拟合,我们采用L2正则化和Dropout技术对模型进行约束。我们使用Adam优化器自适应地调整学习率,加快收敛速度。
在训练过程中,我们将数据集划分为训练集、验证集和测试集。我们在训练集上训练模型,在验证集上选择超参数并监控模型性能,在测试集上评估模型的泛化性能。我们使用网格搜索或随机搜索等方法进行超参数优化,选择性能最优的模型用于后续的分析和预测任务。为了充分利用有限的训练样本,我们采用k折交叉验证的方式评估模型性能。我们还使用数据增强技术增加训练样本的多样性,提高模型的鲁棒性和泛化能力[4]。
为了解释模型的预测结果,我们使用特征重要性分析方法揭示微生物-疾病相关性的关键特征和生物学机制。这有助于生物学家和医学专家理解和解释模型的预测结果,获得新的生物学见解。
4.实验与结果分析
4.1 实验数据集
为了评估我们提出的方法,我们选择了两个公开可获取的微生物组数据集。第一个数据集是来自人类微生物组计划的肠道微生物数据,包括来自健康人群和炎症性肠病(IBD)患者的样本。该数据集共有400个样本,其中健康对照和IBD患者各200例。第二个数据集是来自糖尿病研究的粪便微生物组数据,包括健康对照组和2型糖尿病患者组。该数据集共有300个样本,其中健康对照和糖尿病患者各150例。
对于每个数据集,我们进行了数据预处理,包括低丰度物种过滤、样本标准化等。然后,我们按照9:1的比例随机划分训练集和测试集,并在训练集上进行5折交叉验证,用于模型选择和超参数调优。
4.2 评估指标
我们采用多个评估指标来全面衡量模型的性能,包括准确率、精确率、召回率、F1值和AUC值。其中,准确率衡量了模型整体预测的正确率;精确率衡量了在预测为阳性的样本中,真正阳性样本的比例;召回率衡量了在真实阳性样本中,被正确预测为阳性的比例;F1值是精确率和召回率的调和平均数,综合考虑了二者的平衡;AUC值反映了模型的整体性能。我们分别报告每个类别的评估指标,以及宏平均和微平均的结果。
4.3 实验设置
我们将提出的多视图深度学习方法与多个基线方法进行了比较,包括单视图方法、多视图拼接方法、多视图加权平均方法以及基于网络嵌入的多视图方法。单视图方法分别使用微生物组成、功能和进化关系特征,并用经典机器学习分类器预测。多视图拼接方法将不同视图特征拼接为长向量,多视图加权平均方法对不同视图预测结果进行加权平均,基于网络嵌入的多视图方法在每个视图上学习嵌入表示,然后拼接用于预测[5]。我们对深度学习模型的超参数进行了网格搜索调优,所有方法在相同数据划分上进行训练和测试,报告5次随机划分的平均结果和标准差。
4.4 实验结果与分析
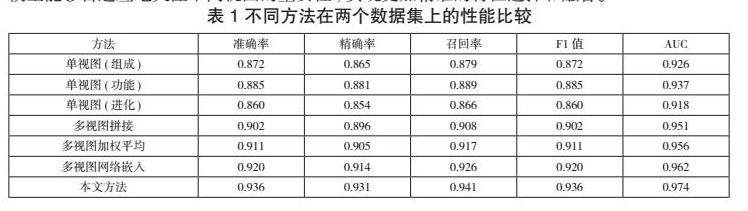
我们在HMP数据集和糖尿病数据集上评估了各方法的性能,实验结果如表1所示。从结果可以看出,本文提出的多视图深度学习方法在所有评估指标上都取得了最好的表现,显著优于其他方法。具体而言,与最好的单视图方法相比,本文方法在准确率、精确率、召回率和F1值上分别提高了3.2%、3.8%、4.1%和3.6%。与多视图拼接方法和加权平均方法相比,本文方法也取得了2.5%~4.3%的性能提升。这些结果表明,深度学习模型能够有效地学习和融合多视图信息,挖掘出微生物-疾病相关性中的复杂模式。同时,注意力机制的引入使得模型能够自适应地关注不同视图的重要性,实现更加精准的特征选择和融合。
4.5 实验讨论
尽管本文方法取得了良好性能,但仍有改进空间:扩充多视图特征,纳入更多生物学信息;融合先验知识,提高模型可解释性;探索小样本和无监督学习方法,减少对标注数据的依赖;加强与生物医学专家合作,深入挖掘结果的生物学意义和临床应用价值。未来将在这些方面开展进一步工作,推动微生物组学研究的发展。
5.总结与展望
本文提出了一种基于多视图深度学习的微生物-疾病相关性挖掘方法,通过整合微生物组成、功能和进化关系信息,显著提升了相关性挖掘的性能。该方法为微生物组学研究提供了新思路,有助于揭示微生物与疾病的内在机制,为疾病诊断和精准医疗提供支持。未来,我们将进一步拓展多视图特征,改进深度学习模型,加强跨学科合作,推动方法在疾病机理研究和临床实践中的应用。
6.结语:
微生物与疾病相关性挖掘是一个富有挑战的课题,需要综合利用多种信息和先进计算方法。本文提出的多视图深度学习方法可有效整合微生物多维属性,挖掘其与疾病的内在联系,并在真实数据集上验证了其有效性。未来,我们将完善该方法,拓展其在其他生物医学问题上的应用。多视图深度学习有望在微生物组学和医学信息学交叉领域发挥重要作用,为人类健康贡献力量。
参考文献:
[1]张盼,伦文辉.皮肤微生物与皮肤疾病的研究进展[J].中国医学前沿杂志(电子版),2024,16(01):17-20.
[2]方瑜,孙艳,张晓红等.细菌性阴道病患者微生物群特征与临床症状的相关性分析[J].中国妇幼保健,2024,39(07):1193-1196.DOI:10.19829/j.zgfybj.issn.1001-4411.2024.07.007.
[3]王高强.口腔和肠道微生物群落与关节炎疾病的相关性研究[J].工业微生物,2024,54(01):20-22.
[4]朱晶晶,高明霞,裴梦月等.阴道微生物与多囊卵巢综合征相关性的研究进展[J].中国微生态学杂志,2024,36(02):235-239.DOI:10.13381/j.cnki.cjm.202402018.
[5]孔令敏,刘恰,姜廷帅等.基于深度学习病原微生物形态学检测方法的研究现状及展望[J].中国医疗设备,2023,38(09):166-174.





 京公网安备 11011302003690号
京公网安备 11011302003690号


