
- 收藏
- 加入书签
基于网络药理学探讨地黄紫草治疗银屑病的有效成分及作用机制
摘要:本文旨在通过网络药理学探讨地黄-紫草药对在银屑病治疗中的活性成分和机制。具体而言,从文献和中药系统药理学数据库(TCMSP)检索了地黄-紫草的化学成分,以及从UniProt获得了这些成分的靶点,还从GeneCards和DisGeNET获得了与银屑病相关的靶点。然后,使用Cytoscape构建了化学成分-药理靶点、蛋白质-蛋白质相互作用(PPI)和化学成分-银屑病靶点网络。基于DAVID数据库进行了基因本体(GO)富集分析和京都基因与基因组百科全书(KEGG)通路富集分析。结果显示,地黄-紫草中有11种化合物,以及在银屑病治疗中的这一药对的19个共同靶点。这19个共同靶点主要涉及生物过程,如干扰素-γ介导的信号通路的负调控(GO: 0060336)、炎症反应的负调控(GO: 0050728)和细胞内受体信号通路(GO: 0030522),以及信号通路,如PPAR信号通路(hsa03320)、化学致癌 - 受体激活(hsa05207)和癌症通路(hsa05200)。根据PPI分析,EGFR可能是银屑病治疗的关键靶点。本研究初步揭示了地黄-紫草药对在银屑病治疗中的多成分、多靶点和多途径机制,为深入研究该药对在银屑病治疗中的机制提供了理论依据。
关键字:网络药理学,银屑病,地黄,紫草,中医。
1.引言
银屑病是皮肤病学中常见的慢性炎症性皮肤疾病,其特点是表皮增厚、免疫反应和血管增生。全球患病率为0.1% ~ 3% [1],而中国的患病率为0.47%,并且中国的银屑病患病率正在上升[2]。现代医学发现,银屑病与多种因素和通路有关,包括遗传、炎症、免疫、微循环紊乱、细胞增殖和凋亡[3-9],其发病机制复杂。中医药可以充分发挥多成分、多靶点和多途径的治疗优势[10-13],中医药研究对改善临床症状、减少疾病复发和提高生活质量非常重要。
中医将银屑病称为"白疕",多因血热、血虚、血瘀引起。历代医家对银屑病的治疗主要以血分为基础。临床上常见的是血热、血燥和血瘀证,证候常交织不清。清热凉血药通常是主要的治疗药物[14],其中,地黄与紫草这一对药使用最为频繁。
在我们的研究中,通过网络药理学推断出地黄-紫草药对在银屑病治疗中的共同靶点,然后使用数据库进行核心靶点的基因本体(GO)功能和京都基因与基因组百科全书(KEGG)通路富集分析,本文在分子水平上预测了地黄-紫草药对治疗银屑病的机制,为进一步的药理学研究和临床试验提供了有效的依据。
材料与方法
地黄与紫草药的活性成分及靶基因筛选
在传统中药数据库和分析平台[17](TCMSP,https://www.tcmspw.com/)上输入中药名字,以获取相应的化学成分及相关信息。根据药代动力学(ADME)原理,以口服生物利用度(OB)≥ 30%和药物相似性(DL)≥0.18作为筛选条件,获得每种药物的有效成分。
搜集与银屑病相关的靶标基因
为确保数据的全面性和准确性,以“银屑病”为关键词在GeneCards [18](https://www.genecards.org/)和DisGeNET [19](https://www.disgenet.org/)中搜索银屑病的潜在靶标。在GeneCards数据库中,分数表示靶标与疾病的关联程度,因此将分数大于中位数的靶标设置为银屑病的潜在靶标。
确定地黄和紫草药在银屑病治疗中的潜在治疗靶标
分别将三个靶标集输入Venny 2.1.0,以获取共同的靶标,并绘制Venn图以获得地黄和紫草药在银屑病治疗中的潜在靶标。
构建药物-化合物-靶标网络
将地黄和紫草的活性成分以及银屑病治疗的潜在靶标导入Cytoscape 3.8.2 [20]软件,绘制药物-化合物-靶标网络图以进行可视化分析,其中“节点”用于表示药物、成分或靶标,“边”用于表示节点之间的关系。使用网络分析器和cytoNCA插件分析每个节点的网络参数。
构建蛋白质-蛋白质相互作用(PPI)网络图和核心靶标筛选
将上述筛选得到的潜在靶标导入STRING [21](https://www.string-db.org/),选择“Homo sapiens”,将最低要求的相互作用分数设置为≥ 0.4,隐藏自由节点,构建PPI网络,并导出相应的文件。下载的结果上传到Cytoscape 3.8.2软件进行分析。通过以度的两倍中值,以及介数中心性(BC)和接近中心性(CC)的中值作为卡值,筛选出核心靶标。
基因本体(GO)功能富集和京都基因与基因组百科全书(KEGG)通路分析
将先前获得的潜在靶标导入DAVID数据库 [22](https://david.ncifcrf.gov/),物种定义为“Homo sapiens”。分析地黄和紫草药的共同靶标基因以及银屑病的GO功能和KEGG通路富集。使用生物信息学在线绘图工具(http://www.bioinformatics.com.cn/)可视化结果,并使用Cytoscape 3.8.2构建“疾病-通路-靶标-成分药物”的网络图。
分子对接
在PDB数据库中搜索核心基因以进行分子对接实验,下载相应蛋白质的结构[23]。使用PyMOL(版本2.4.0)软件从这些受体大分子结构中去除水分子和小配体分子[24]。使用AutoDockTools(版本1.5.6)软件添加极性氢离子[25],并设置网格框以确定蛋白质的活性口袋的搜索范围,这些口袋是未知的。为了包含尽可能多的受体结构,必须适度增加活性口袋的大小以提高对接结果。根据已识别的核心基因,寻找与核心基因相互作用的药物成分,这些小配体分子的二维结构从PubChem数据库下载,使用ChemBio3D(版本14.0)软件中的MM2计算工具优化具有最小自由能的小配体分子的三维结构。使用Vina(版本1.1.2)软件进行分子对接工作,并计算最佳的分子对接模型。
结果
活性成分和HLXLD的靶标筛选
通过TCMSP数据库检索,地黄和紫草分别含有76种和51种活性成分。在地黄药材的所有活性成分中,排名前六位的分别是酪氨酸葡聚糖、β-谷甾醇、胶苷、地黄甙A、6-O-香草酰阿举固醇和γ-谷甾醇;在紫草药材的所有活性成分中,排名前六位的分别是曼得醇、(6Z,10E,14E,18E)-2,6,10,15,19,23-六甲基二十四碳二烯、油酸乙酯 (NF)、β-谷甾醇、乌头酮和去-O-甲基拉西奥地普二烯。合并并去除重复后,获得了总共11种活性成分,并进一步获取了相应的靶蛋白。在转化为UniProt数据库中的标准基因名称并去除重复后,总共获得了340个预测靶标。
银屑病相关靶标蛋白的收集
在合并和去重GeneCards和DisGeNET数据库收集的银屑病疾病靶标基因后,获得了总共1529个相关的靶标蛋白。将地黄和紫草的活性成分的靶标基因与与银屑病相关的靶标基因进行匹配,取交集,通过Venn图获得了19个共同基因,19个共同基因的信息如表1所示。
药物-化合物-靶标网络的构建
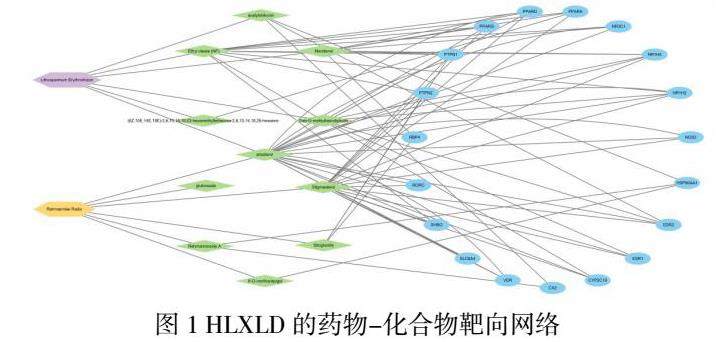
引入地黄和紫草在银屑病治疗中的活性成分及其相应的靶标进入Cytoscape 3.10.0软件中,构建包含32个节点和78个边的药物-化合物-靶标网络图(图1)。其中,紫色六边形节点代表紫草,黄色六边形节点代表地黄,绿色菱形节点代表地黄和紫草药的有效成分,蓝色椭圆节点代表药物和疾病的共同靶标。
PPI网络构建和拓扑分析
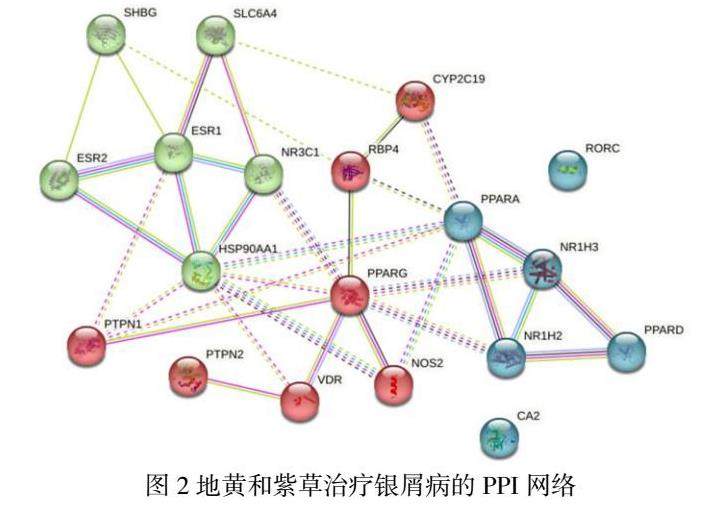
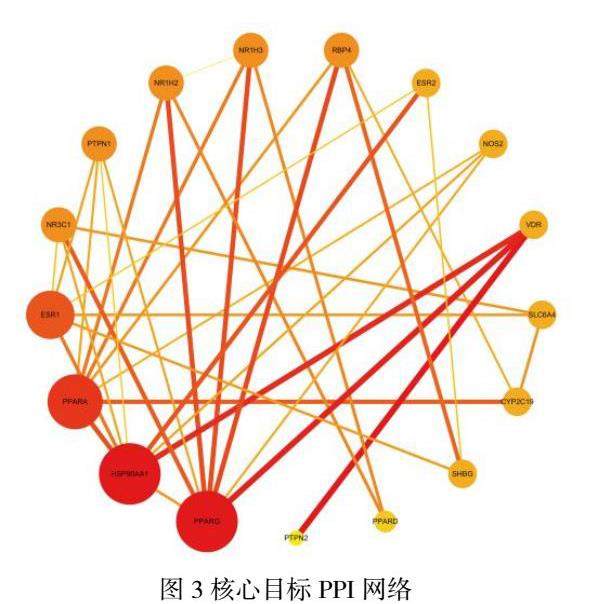
将19个交集靶标引入STRING数据库以建立PPI网络(图2)。该网络由17个节点和35个边组成。这些靶标在PPI网络中起关键作用,是地黄和紫草在治疗银屑病中的核心靶标。导入Cytoscape 3.10.0软件,进一步可视化核心蛋白质相互作用(图3)。节点的大小和颜色反映了度的大小。节点越大,红色越浓表示度越高。边的粗细反映了节点之间连接分数的大小。颜色越浓表示节点之间连接分数越大。
KEGG通路富集分析
通过KEGG通路富集分析共获得7个通路。根据P值绘制气泡图。通过Cytoscape 3.10.0进一步构建疾病-通路-靶标-成分药物网络。该网络包括40个节点(包括1个疾病、7个通路、19个靶标、11个成分和2个药物)和112个边。其中,紫色六边形节点代表紫草,黄色六边形节点代表地黄,绿色菱形节点代表地黄和紫草药的有效成分,蓝色椭圆节点代表药物和疾病的共同靶标,红色三角形节点代表KEGG通路,灰色矩形节点代表疾病。
主要活性成分与核心靶标的分子对接
从PPI核心网络中选择了两个核心靶标:PPARG和HSP90AA1。从PDB数据库中检索了PPARG和HSP90AA1蛋白质。我们对受体蛋白质大分子进行了预处理,去除了水分子和小配体分子,添加了极性氢,然后设置了活性口袋的搜索范围。根据药物-化合物-靶标网络,选择了与核心靶标相互作用的活性成分:Mandenol、Ethyl oleate (NF)、Sitosterol、Stigmasterol(用于PPARG)和Acetylshikonin、Rehmannioside A、6-O-vanilloylajugol(用于HSP90AA1)。
我们使用Vina软件验证了受体大分子与中药小分子成分之间的分子对接。结果如表3所示。我们选择了结合能最小的模型,并使用PyMOL软件进行可视化。结合能越小,配体与受体之间的结合越强。当亲和力小于-4.25 kcal/mol时,配体与受体显示出一定的结合性。亲和力小于-5.0 kcal/mol表示良好的结合活性,而亲和力小于-7.0 kcal/mol表示强烈的结合活性。Stigmasterol-PPARG和Acetylshikonin-HSP90AA1具有最高的亲和力。
讨论
银屑病是一种病程长且容易复发的皮肤病,其发病机制尚未完全阐明。对于银屑病的现代医学治疗方法的长期使用仍然具有挑战性。中药制剂是复杂的药物体系。它们的多种成分和多靶点之间的复杂相互作用可以导致对各种复杂疾病的调控,从而为治疗提供可能。中药治疗银屑病的疗效不亚于现代医学疗法的疗效;此外,它可能加强现代医学疗法的疗效。由于其在临床应用方面的独特理论和成功用于银屑病的治疗,我们认为基于中药开发新的小分子药物将是为银屑病患者提供安全、有效且成本较低的治疗的有前途的策略。因此,我们采用了网络药理学研究方法,从活性成分、靶基因和信号通路的角度探讨了地黄和紫草如何治疗银屑病的机制。
结果显示,在地黄和紫草治疗银屑病中存在19个潜在靶点,以及涉及银屑病治疗的11种主要活性成分,如豆甾醇和乙酰紫草素。 豆甾醇存在于大豆油和其他大豆油中,有时独立存在,有时以甘苷的形式存在。甾醇具有高营养价值,它具有抗炎、抗氧化、促进皮肤代谢和抑制皮肤炎症的作用,还可以抑制胆固醇的吸收,加速体内胆固醇的代谢。
地黄和紫草治疗银屑病的潜在靶点PPI网络表明,地黄和紫草可能通过作用于PPARG和HSP90AA1等核心靶点来治疗银屑病。许多其他研究也报告了PPARG蛋白表达水平与银屑病发病率之间的强相关性。
GO功能富集分析和KEGG通路富集分析的结果表明,地黄和紫草治疗银屑病的机制主要集中在胆固醇储存的负调控、从RNA聚合酶II启动子的转录调控、干扰素-γ介导的信号通路的负调控、激素介导的信号通路、从RNA聚合酶II启动子的pri-miRNA转录的负调控、正调控转录、细胞内受体信号通路、炎症反应的负调控等多个方面,而地黄和紫草治疗银屑病的相关通路主要涉及PPAR信号通路、化学致癌物 - 受体激活、癌症通路、雌激素信号通路等。
在本研究中,我们使用网络药理学建立了药物靶点通路和网络,并预测了地黄和紫草治疗银屑病的可能蛋白质靶点以及这些靶点与药物之间的主要相互作用。然而,该研究缺乏实验验证,但它提供了进一步进行体外和体内实验研究以探索地黄和紫草与银屑病之间详细机制的可能方向。
参考文献:
[1]Danielsen K, Olsen AO, Wilsgaard T, Furberg AS. Is the prevalence of psoriasis increasing? A 30-year follow-up of a population-based cohort. Br J Dermatol. 2013. 168(6): 1303-10.
[2]Li J, Yu M, Wang YW, et al. Prevalence of psoriasis and associated risk factors in China: protocol of a nationwide, population-based, cross-sectional study. BMJ Open. 2019. 9(7): e027685.
[3]Kunz M, Simon JC, Saalbach A. Psoriasis: Obesity and Fatty Acids. Front Immunol. 2019. 10: 1807.
[4]Das D, Akhtar S, Kurra S, Gupta S, Sharma A. Emerging role of immune cell network in autoimmune skin disorders: An update on pemphigus, vitiligo and psoriasis. Cytokine Growth Factor Rev. 2019. 45: 35-44.
[5]周婧,徐鹏,杨波,柏冰雪,孙丽艳,孙东林.ADAM33基因多态性与东北汉族银屑病的相关性[J].中国麻风皮肤病杂志,2014,(9):530-534.
[6]Yao X, Hao S, Yu P. Association Study of the Caspase Gene Family and Psoriasis Vulgaris Susceptibility in Northeastern China. Biomed Res Int. 2019. 2019: 2417612.
[7]Fan X, Wang H, Sun L, et al. Fine mapping and subphenotyping implicates ADRA1B gene variants in psoriasis susceptibility in a Chinese population. Epigenomics. 2019. 11(4): 455-467.
[8]Luo Q, Zeng J, Li W, et al. Interaction of MTHFR gene with smoking and alcohol use and haplotype combination susceptibility to psoriasis in Chinese population. Immunol Res. 2018. 66(4): 543-547.
[9]Lorscheid S, Müller A, Löffler J, et al. Keratinocyte-derived IκBζ drives psoriasis and associated systemic inflammation. JCI Insight. 2019. 4(22): e130835.
[10]Meng S, Lin Z, Wang Y, Wang Z, Li P, Zheng Y. Psoriasis therapy by Chinese medicine and modern agents. Chin Med. 2018. 13: 16.
[11]Deng J, Yao D, Lu C, et al. Oral Chinese herbal medicine for psoriasis vulgaris: protocol for a randomised, double-blind, double-dummy, multicentre clinical trial. BMJ Open. 2017. 7(11): e014475.
[12]Chiang CC, Cheng WJ, Lin CY, et al. Kan-Lu-Hsiao-Tu-Tan, a traditional Chinese medicine formula, inhibits human neutrophil activation and ameliorates imiquimod-induced psoriasis-like skin inflammation. J Ethnopharmacol. 2020. 246: 112246.
[13]Xu M, Deng J, Xu K, et al. In-depth serum proteomics reveals biomarkers of psoriasis severity and response to traditional Chinese medicine. Theranostics. 2019. 9(9): 2475-2488.
[14]Chen X, Zhang R, Duan X, Xue M, Qu T, Li L. Effectiveness of Xiaoyin Jiedu granules in the treatment of psoriasis vulgaris in patients with blood-heat symptom patterns in terms of Traditional Chinese Medicine. J Tradit Chin Med. 2020. 40(5): 863-869
[17]Ru J, Li P, Wang J, et al. TCMSP: a database of systems pharmacology for drug discovery from herbal medicines. J Cheminform. 2014. 6: 13.
[18]Stelzer G, Rosen N, Plaschkes I, et al. The GeneCards Suite: From Gene Data Mining to Disease Genome Sequence Analyses. Curr Protoc Bioinformatics. 2016. 54: 1.30.1-1.30.33.
[19]Piñero J, Ramírez-Anguita JM, Saüch-Pitarch J, et al. The DisGeNET knowledge platform for disease genomics: 2019 update. Nucleic Acids Res. 2020. 48(D1): D845-D855.
[20]Ragueneau E, Shrivastava A, Morris JH, Del-Toro N, Hermjakob H, Porras P. IntAct App: a Cytoscape application for molecular interaction network visualization and analysis. Bioinformatics. 2021. 37(20): 3684-3685.
[21]Szklarczyk D, Gable AL, Nastou KC, et al. The STRING database in 2021: customizable protein-protein networks, and functional characterization of user-uploaded gene/measurement sets. Nucleic Acids Res. 2021. 49(D1): D605-D612.
[22]Huang da W, Sherman BT, Lempicki RA. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nat Protoc. 2009. 4(1): 44-57.
[23]Berman HM, Westbrook J, Feng Z, et al. The Protein Data Bank. Nucleic Acids Res. 2000. 28(1): 235-42.
[24]Mooers B. Shortcuts for faster image creation in PyMOL. Protein Sci. 2020. 29(1): 268-276.
[25]Trott O, Olson AJ. AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization, and multithreading. J Comput Chem. 2010. 31(2): 455-61.
基金项目:1.邢台市重点研发计划自筹项目(2021ZC120)





 京公网安备 11011302003690号
京公网安备 11011302003690号


